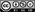RESUMEN
Una vez establecida la sospecha clínica y radiológica, el diagnóstico de certeza de una neoplasia cerebral requiere el estudio directo del tejido tumoral obtenido mediante biopsia o resección quirúrgica.
En la actualidad, el diagnóstico de los tumores cerebrales se basa en los criterios revisados de la Organización Mundial de la Salud, OMS (Louis et al; 2007). Su fundamento es el estudio histológico apoyado por la inmunohistoquímica, pero en los últimos años se están integrando hallazgos moleculares relevantes, tanto en investigación como en la práctica clínica especializada. Actualmente, el estudio del tejido tumoral debe integrar técnicas histológicas, inmunohistoquímicas y de genética molecular (Hunt; 2008, Pfeifer; 2010).
El objetivo del empleo de estas técnicas no solo es confirmar el diagnóstico de un tumor y establecer su naturaleza, sino también dar información pronóstica e incluso predictiva de respuesta al tratamiento, como en el caso de la codelección 1p 19q en oligodendrogliomas, o la metilación del promotor de la enzima reparadora de ADN O-6-metil-guanina-ADN-metil-transferasa (MGMT) en glioblastomas.
En este capítulo se mencionarán las principales técnicas histopatológicas, inmunohistoquímicas, citogenéticas y de genética molecular, así como su aplicación al diagnóstico de tumores cerebrales. Se profundiza en el diagnóstico molecular de los gliomas. Se incluyen recursos web en un apéndice.
TÉCNICAS DE ESTUDIO
En el apéndice se incluyen enlaces que contienen explicaciones esquemáticas de muchas de las técnicas.
- Histopatología
Comprende la valoración macro y microscópica del tejido tumoral. Informa de la presencia de tumor, márgenes, origen neurológico o no, clasificación histológica y grado. La heterogeneidad tumoral obliga a estudiar múltiples fragmentos. Se debe incluir información intraoperatoria que ayude en la decisión quirúrgica (aproximación diagnóstica y bordes tumorales).
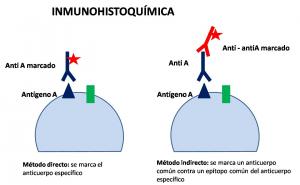
- Inmunohistoquímica
Es imprescindible en el diagnóstico patológico (Jaffer et al; 2004). Se basa en el reconocimiento de antígenos tisulares por anticuerpos específicos. Ha progresado enormemente con el desarrollo de anticuerpos monoclonales de alta especificidad. [inmunohistoquímica.jpg]
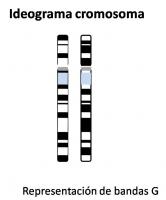
- Citogenética
Valora alteraciones cromosómicas. Las técnicas fundamentales son:
- Citogenética convencional, de bandas. [bandasg.jpg]
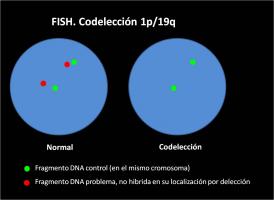
- Citogenética molecular: Se basa en técnicas de hibridación in situ con fluorescencia (FISH). [FISH.jpg]
Localiza fragmentos de ADN en su localización cromosómica. Las nuevas técnicas que combinan microarrays (hibridación genómica comparativa –CGH-, etc.), permiten estudios a mucha menor escala (Espinet et al; 2005).
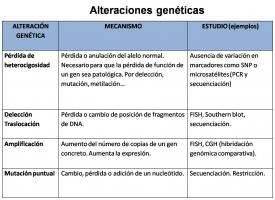
- Genética molecular
Valora alteraciones genéticas a nivel molecular. Estas técnicas están permitiendo profundizar en los mecanismos patogénicos del cáncer, tanto en las alteraciones que se producen en genes relacionados con la oncogénesis [alteraciones genéticas.jpg]
como en los mecanismos epigenéticos que controlan su expresión [epigenética.jpg].
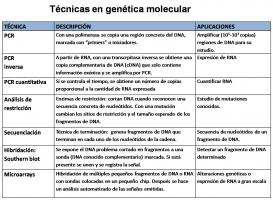
Se mencionan alguna de las técnicas más importantes (Jiménez Escrig; 2007): [técnicas genética molecular.jpg]
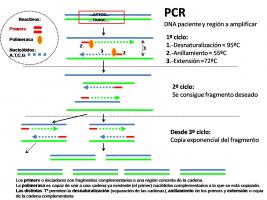
- PCR, reacción en cadena de la polimerasa: [PCR.jpg]
Amplifica regiones de ADN. Para amplificar ARN y cuantificar su expresión se emplea PCR inversa y PCR cuantitativa.
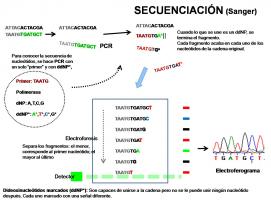
- Secuenciación: [secuenciación.jpg]
Determina la secuencia de nucleótidos de una región de ADN.
- Técnicas indirectas para estudio de mutaciones como el análisis de restricción. [restricción.jpg]
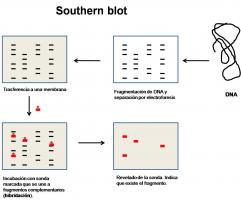
- Hibridación: Valora la presencia de fragmentos con una secuencia de nucleótidos concreta. Incluye las técnicas de Southern blot (ADN) [southern blot.jpg], Northern blot (ARN).
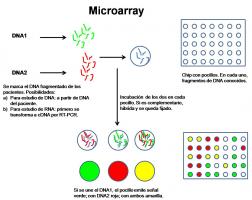
- Microarrays (chip de nucleótidos): [microarray.jpg]
Basada en la anterior, incorpora técnicas automatizadas de alto rendimiento. Estudia numerosos fragmentos de ADN y ARN en un pequeño chip al mismo tiempo, lo que ha supuesto una auténtica revolución en genética molecular (Shen et al; 2009).
APLICACIÓN A LOS TUMORES CEREBRALES
En la actualidad, el diagnóstico de los tumores cerebrales se basa en los criterios revisados de la OMS (Louis et al; 2007). Su fundamento sigue siendo el estudio histológico apoyado por la inmunohistoquímica, pero en los últimos años se están integrando hallazgos moleculares relevantes, tanto en investigación como en la práctica clínica especializada.
El estudio histológico se centra en: 1) aspectos morfológicos en función de su posible histogénesis (tumores astrocíticos, oligodendrogliales, neuronales, etc.); 2) grado tumoral (I y II bajo grado; III y IV alto grado) en función de la presencia o no de alteraciones como atipia nuclear, mitosis, necrosis y proliferación vascular. Estos dos últimos rasgos, además de clasificar los tumores, constituyen el principal factor pronóstico conocido en la actualidad.
La inmunohistoquímica contribuye a diagnosticar la estirpe tumoral (proteína S-100, proteína gliofibrilar acídica…), su grado y pronóstico (marcador de proliferación celular Ki-67, proteína p53…). La presencia de células CD133+, indica un tumor de células progenitoras de muy mal pronóstico (Singh et al; 2003).
Las técnicas moleculares no solo tiene aplicación en investigación sino también en el diagnóstico, ayudando a discriminar grupos de diferente pronóstico y de diferente respuesta a los tratamientos (Huse et al; 2010).
PATOLOGÍA MOLECULAR DE LOS GLIOMAS
Los estudios anatomopatológicos tradicionales no permiten establecer un pronóstico ajustado en muchos casos. Por ejemplo, qué gliomas de bajo grado progresarán a alto grado; qué significación tienen variantes del glioblastoma como el de células gigantes; cómo diferenciar de forma fiable un glioblastoma primario de uno secundario, etc.
Se está profundizando en el conocimiento de las principales vías implicadas en la patogénesis de los gliomas como la de la proteína p53, la del gen supresor tumoral del retinoblastoma o las relacionadas con los receptores tirosín-quinasa (Ras-MAPK; AKT-mTOR…). Se pueden encontrar esquemas de estas vías en la página del KEGG (
http://www.genome.jp/kegg/pathway/hsa/hsa05214.html) y otros artículos (Cancer Genome Atlas Research Network; 2008)(Huse et al; 2010).
Estos nuevos conocimientos han permitido iniciar el estudio de marcadores moleculares de importancia pronóstica y predictiva en gliomas, y plantear posibles clasificaciones patomoleculares.
- Marcadores moleculares (Ducray et al; 2009).
Constantemente se plantean nuevos factores moleculares (Sheng et al; 2010), cuya trascendencia habrá que ir validando en sucesivos estudios. Los mejor caracterizados en la actualidad son: [marcadores glioma.jpg]
-Metilación del gen reparador del ADN, O-6-metil-guanina-ADN-metil-transferasa (MGMT): Es un factor de gran valor en la respuesta a tratamiento en glioblastomas. Indica buen pronóstico (Hegi et al; 2005). Si la región promotora del gen MGMT está metilada, el gen no se expresa y la célula es más sensible a la quimio-radioterapia porque el DNA dañado se repara menos eficazmente. Se ha visto que el estado de metilación puede cambiar con el tiempo (Brandes et al; 2010). Dada su importancia, se está incorporando a estudios rutinarios, siendo necesaria la estandarización de las técnicas empleadas (Weller et al; 2010). Se están buscando nuevas posibilidades, como su determinación en fluidos en lugar del tejido tumoral (Lavon et al; 2010).
- Codelección 1p/19q [FISH.jpg]: En oligodendrogliomas, se asocia a mayor supervivencia y respuesta a tratamiento (Cairncross et al; 1998). Están en discusión: el mecanismo patogénico, que podría relacionarse con IDH1/IDH2 (Labussière et al; 2010); el papel en astrocitomas (Vogazianou et al; 2010); y la mejor técnica para su estudio.
- Mutaciones en IDH-1/IDH-2: Están presentes en gliomas II y III y en glioblastomas secundarios, pero no en los primarios (Parsons et al; 2008, Yan et al; 2009). Por tanto, indicaría mejor pronóstico en glioblastomas. También podría emplearse como marcador diagnóstico frente a los astrocitomas pilocíticos (Korshunov et al; 2009). Inducen oncogénesis por la vía HIF-1 (Zhao et al; 2009).
- Mutaciones en el gen TP53: Las alteraciones en la proteína p53 favorecen la oncogénesis al alterarse sus múltiples funciones (control transcripcional, apoptosis, ciclo celular…). Se han implicado en la progresión de gliomas de bajo grado a glioblastoma (Nozaki et al; 1999).
- Amplificación y mutaciones del gen del receptor del factor de crecimiento epidérmico EGFR (Libermann et al; 1985): Se relaciona con glioblastomas primarios, implicando mal pronóstico. La sobreexpresión o la mutación EGFRvIII, muy frecuente en glioblastomas (Gan et al; 2009), dan lugar a hiperfunción de este receptor tirosín-quinasa, por lo que se ha planteado el tratamiento con inhibidores específicos como el erlotinib. Sin embargo, solo una minoría de tumores responden: serían los pacientes que tienen la mutación EGRFvIII y no tienen alteraciones en PTEN (Mellinghoff et al; 2005, Lo; 2010).
- Hacia una clasificación molecular de los gliomas.
Con el análisis masivo de expresión de ARN, se están estudiando perfiles de expresión, que valoran conjuntos de factores más que marcadores aislados, con un alto valor predictivo (Castells et al; 2010, Gravendeel et al; 2009).
El análisis de expresión de ARN en gliomas junto con estudios genéticos y de proteínas, está llevando a plantear clasificaciones moleculares, especialmente en glioblastomas (Brennan et al; 2009), Verhaak et al; 2010). Se trataría de entidades diferentes con una biología molecular particular, más que variantes de una misma entidad. Recientemente se ha descrito un fenotipo hipermetilador que se asocia a mejor pronóstico (Noushmehr et al; 2010).
PERSPECTIVAS FUTURAS
Se deben incorporar técnicas de biología molecular al diagnóstico de rutina histopatológico e inmunohistoquímico de los tumores cerebrales. Actualmente es imprescindible, al menos, el estudio de la metilación de MGMT en glioblastomas y la codelección 1p/19q en oligodendrogliomas.
Es necesario emplear métodos sencillos y estandarizados. Esto exige la adopción de criterios de validación universales (Jennings et al; 2009). Además, se están desarrollando técnicas para estudiar en sangre periférica estos marcadores genéticos (metilación de MGMT, etc.) (Lavon et al; 2010).
En investigación, se están realizando estudios a gran escala empleando nuevas tecnologías como microarrays (Parsons et al; 2008). Permiten profundizar en el análisis de vías patogénicas, nuevos marcadores moleculares, perfiles de expresión de ARN, el papel de otros mecanismos epigenéticos como los micro-ARN (Turner et al; 2010) o el desarrollo de nuevas estrategias terapéuticas basadas en la biología molecular del cáncer (Yamanaka et al; 2009).
Debe destacarse la necesidad de estudios multicéntricos y el papel de redes de estudio de genética del cáncer como
The Cancer Genome Atlas Research Network (
http://cancergenome.nih.gov/)(Cancer Genome Atlas Research Network; 2008).
RECURSOS WEB
BASES DE DATOS CON ENLACES CON OTROS RECURSOS DE BIOLOGÍA MOLECULAR:
-CDC (Centers for Disease Control and Prevention): Genomics resources.
-AACC (American Association for Clinical Chemistry): Molecular Pathology Division Related Links.
MÉTODOS (PÁGINAS DIVULGATIVAS):
-Doland DNA Learning Center. Resources. Biology Animation Library y 3-D Animation Library.
Solo algunas de las técnicas fundamentales. Nivel divulgativo, muy claro.
-Campbell AM, Heyer LJ. Discovering genomics, proteomics and bioinformatics.
El acceso es algo farragoso pero es muy completo. Nivel divulgativo, muy claro.
GENÉTICA GENERAL:
-OMIM: Online Mendelian Inheritance in Man. NCBI.
Revisión bibliográfica de todos los trastornos genéticos conocidos, haciendo hincapié en la relación entre fenotipo y genotipo.
-GENECARDS: The Human Gene Compendium.
Genes, estructura, transcripción, proteínas, funciones, patología, bibliografía…
-ENSEMBL.
Información del genoma de vertebrados y otros eucariotas.
-KEGG: Kyoto Encyclopedia of Genes and Genomes.
Genes y sus implicaciones biológicas, desde su estructura básica a vías patogénicas de enfermedades.
INMUNOHISTOQUÍMICA:
-Immunohistochemistry vade mecum.
Inmunohistoquímica de los tumores y principales antígenos estudiados.
CITOGENÉTICA:
-Cytogenetic resources.
Enlaces con recursos en la web en citogenética.
PATOLOGÍA MOLECULAR EN CÁNCER:
-CNIO: Centro Nacional de Investigaciones Oncológicas.
Centro para el diagnóstico y la investigación molecular del cáncer en España.
-COSMIC: Catalogue of somatic mutations in cancer.
Base con las mutaciones somáticas descritas en cáncer de los distintos tejidos.
-ATLAS: The Atlas of Genetics and Cytogenetics in Oncology and Hematology.
alteraciones genéticas en cáncer. Incluye revisiones, tutoriales y enlaces con publicaciones y bases de datos.
-TCGA: The cáncer genome atlas research network.
Red integrada de centros y recursos para el estudio de las bases moleculares de los tumores a gran escala. Citado en bibliografía.
BIBLIOGRAFÍA
Brandes AA, Franceschi E, Tosoni A, Bartolini S, Bacci A, Agati R, et al. (2010). O(6)-methylguanine DNA-methyltransferase methylation status can change between first surgery for newly diagnosed glioblastoma and second surgery for recurrence: clinical implications. Neuro Oncol. 12: 283-288.
Brennan C, Momota H, Hambardzumyan D, Ozawa T, Tandon A, Pedraza A, et al. (2009). Glioblastoma subclasses can be defined by activity among signal transduction pathways and associated genomic alterations. PLoS One. 4: e7752.
Cairncross JG, Ueki K, Zlatescu MC, Lisle DK, Finkelstein DM, Hammond RR, et al. (1998). Specific genetic predictors of chemotherapeutic response and survival in patients with anaplastic oligodendrogliomas. J Natl Cancer Inst. 90: 1473-1479.
Cancer Genome Atlas Research Network. (2008). Comprehensive genomic characterization defines human glioblastoma genes and core pathways. Nature. 23: 1061-1068.
Castells X, Acebes JJ, Boluda S, Moreno-Torres A, Pujol J, Julià-Sapé M,et al. (2010). Development of a predictor for human brain tumors based on gene expression values obtained from two types of microarray technologies. OMICS. 14: 157-164.
Ducray F, El Hallani S, Idbaih A. (2009). Diagnostic and prognostic markers in gliomas. Curr Opin Oncol. 21: 537-542.
Espinet B, Salido M, Solé F. Técnicas de citogenética molecular y sus aplicaciones. Congreso SEAP 2005. Acceso el 20 de junio de 2010. http://www.seapcongresos.com/2005/Cursos/Curso_Largo_Patologia_Molecular/Citogenetica_molecular.PDF
Gan HK, Kaye AH, Luwor RB. (2009). The EGFRvIII variant in glioblastoma multiforme. J Clin Neurosci. 16: 748-754.
Gravendeel LA, Kouwenhoven MC, Gevaert O, de Rooi JJ, Stubbs AP, Duijm JE, et al. (2009). Intrinsic gene expression profiles of gliomas are a better predictor of survival than histology. Cancer Res. 69: 9065-9072.
Hegi ME, Diserens AC, Gorlia T, Hamou MF, de Tribolet N, Weller M, et al. (2005). MGMT gene silencing and benefit from temozolomide in glioblastoma. N Engl J Med. 352: 997-1003.
Hunt JL. (2008). Molecular pathology in anatomic pathology practice: a review of basic principles. Arch Pathol Lab Med. 132: 248-260.
Huse JT, Holland EC. (2010). Targeting brain cancer: advances in the molecular pathology of malignant glioma and medulloblastoma. Nat Rev Cancer. 10: 319-331.
Jaffer S, Bleiweiss IJ. (2004). Beyond hematoxylin and eosin--the role of immunohistochemistry in surgical pathology. Cancer Invest. 22: 445-465.
Jennings L, Van Deerlin VM, Gulley ML; College of American Pathologists Molecular Pathology Resource Committee. (2009). Recommended principles and practices for validating clinical molecular pathology tests. Arch Pathol Lab Med. 133: 743-755.
Jiménez Escrig A. (2007). Molecular biology techniques in investigation and diagnosis. En: Jiménez Escrig A (Ed). Textbook of neurogenetics, (pp 23-37). Madrid: Díaz de Santos.
KEGG. Pathways: Glioma – Homo sapiens. Acceso el 22 de junio de 2010. http://www.genome.jp/kegg/pathway/hsa/hsa05214.html
Korshunov A, Meyer J, Capper D, Christians A, Remke M, Witt H, et al. (2009). Combined molecular analysis of BRAF and IDH1 distinguishes pilocytic astrocytoma from diffuse astrocytoma. Acta Neuropathol. 118: 401-405.
Labussière M, Idbaih A, Wang XW, Marie Y, Boisselier B, Falet C, et al. (2010). All the 1p19q codeleted gliomas are mutated on IDH1 or IDH2. Neurology. 74: 1886-1890.
Lavon I, Refael M, Zelikovitch B, Shalom E, Siegal T. (2010). Serum DNA can define tumor-specific genetic and epigenetic markers in gliomas of various grades. Neuro Oncol. 12: 173-180.
Libermann TA, Nusbaum HR, Razon N, Kris R, Lax I, Soreq H, et al. (1985). Amplification, enhanced expression and possible rearrangement of EGF receptor gene in primary human brain tumours of glial origin. Nature. 313: 144-147.
Lo HW. (2010). EGFR-targeted therapy in malignant glioma: novel aspects and mechanisms of drug resistance. Curr Mol Pharmacol. 3:37-52.
Louis DN, Ohgaki H, Wiestler OD, Cavenee WK, Burger PC, Jouvet A, et al. (2007). The 2007 WHO classification of tumours of the central nervous system. Acta Neuropathol. 114: 97-109.
Mellinghoff IK, Wang MY, Vivanco I, Haas-Kogan DA, Zhu S, Dia EQ, et al. (2006). Molecular determinants of the response of glioblastomas
to EGFR kinase inhibitors. N Engl J Med. 353: 2012-2024.
Noushmehr H, Weisenberger DJ, Diefes K, Phillips HS, Pujara K, Berman BP, et al. (2010). Identification of a CpG island methylator phenotype that defines a distinct subgroup of glioma. Cancer Cell. 17:510-522.
Nozaki M, Tada M, Kobayashi H, Zhang CL, Sawamura Y, Abe H, et al. (1999). Roles of the functional loss of p53 and other genes in astrocytoma tumorigenesis and progression. Neuro Oncol. 1:124-37.
Parsons DW, Jones S, Zhang X, Lin JC, Leary RJ, Angenendt P, et al. (2008). An integrated genomic analysis of human glioblastoma multiforme. Science. 321: 1807-1812.
Pfeifer JD. (2010). Molecular pathology and patient care. Mo Med. 107: 86-93.
Singh SK, Clarke ID, Terasaki M, Bonn VE, Hawkins C, Squire J, et al. (2003). Identification of a cancer stem cell in human brain tumors. Cancer Res. 63: 5821-5828.
Shen Y, Wu BL. (2009). Microarray-based genomic DNA profiling technologies in clinical molecular diagnostics. Clin Chem. 55: 659-669.
Sheng Z, Li L, Zhu LJ, Smith TW, Demers A, Ross AH, et al. (2010). A genome-wide RNA interference screen reveals an essential CREB3L2-ATF5-MCL1 survival pathway in malignant glioma with therapeutic implications. Nat Med. 16: 671-617.
The Cancer Genome Atlas Research Network. Acceso el 20 de junio de 2010. http://cancergenome.nih.gov/
Turner JD, Williamson R, Almefty KK, Nakaji P, Porter R, Tse V, et al. (2010). The many roles of microRNAs in brain tumor biology. Neurosurg Focus. 28: E3.
Verhaak RG, Hoadley KA, Purdom E, Wang V, Qi Y, Wilkerson MD, et al. (2010). Integrated genomic analysis identifies clinically relevant subtypes of glioblastoma characterized by abnormalities in PDGFRA, IDH1, EGFR, and NF1. Cancer Cell. 17: 98-110.
Vogazianou AP, Chan R, Bäcklund LM, Pearson DM, Liu L, Langford CF, et al. (2010). Distinct patterns of 1p and 19q alterations identify subtypes of human gliomas that have different prognoses. Neuro Oncol. Epub 2010 Feb 17.
Weller M, Stupp R, Reifenberger G, Brandes AA, van den Bent MJ, Wick W, et al. (2010). MGMT promoter methylation in malignant gliomas: ready for personalized medicine? Nat Rev Neurol. 6: 39-51.
Yamanaka R, Saya H. (2009). Molecularly targeted therapies for glioma. Ann Neurol. 66: 717-729.
Yan H, Parsons DW, Jin G, McLendon R, Rasheed BA, Yuan W, et al. (2009). IDH1 and IDH2 mutations in gliomas. N Engl J Med. 360: 765-773.
Zhao S, Lin Y, Xu W, Jiang W, Zha Z, Wang P, et al. (2009). Glioma-derived mutations in IDH1 dominantly inhibit IDH1 catalytic activity and induce HIF-1alpha. Science. 324: 261-265.